GLICOPROTEÍNA
Fullføringsbetingelser
Glicoproteína
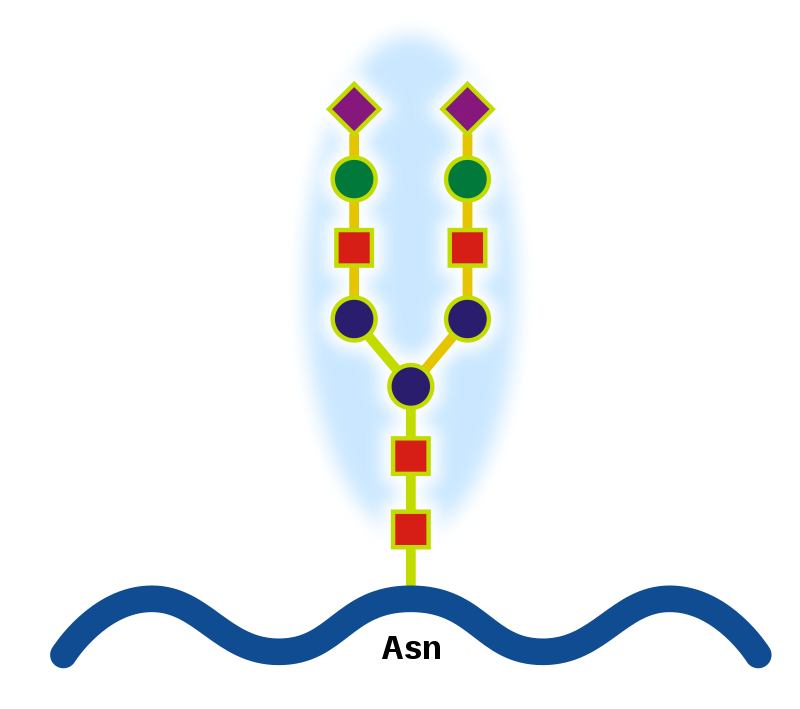
Las glucoproteínas o glicoproteínas son moléculas compuestas por una proteína unida a uno o varios glúcidos, simples o compuestos.
(diseñado por Kosi Gramatikoff)
Las glucoproteínas o glicoproteínas son moléculas compuestas por una proteína unida a uno o varios glúcidos, simples o compuestos. Destacan entre otras funciones la estructural y el reconocimiento celular cuando están presentes en la superficie de las membranas plasmáticas (glucocálix).
El término se usa en general para referirse a una molécula de dimensiones específicas, integrada normalmente por uno o más oligosacáridos unidos de modo covalente a cadenas laterales específicas de polipéptidos. Suelen tener un mayor porcentaje de proteínas que de glúcidos. Los términos proteoglicano y peptidoglicano designan agregados masivos formados por glúcidos y proteínas o séptimos péptidos, para los cuales la palabra molécula no tiene significado preciso. Las partículas de proteoglicanos tienen un mayor porcentaje de glúcidos que de proteínas.
( imagen De Yikrazuul )
La estructura 3D de la glicoproteína espicular del coronavirus SARS-CoV-2.
Francisco R. Villatoro, el 24 febrero, 2020.

Desde que se publicó el genoma del betacoronavirus SARS-CoV-2 he deseado ver la imagen tridimensional de su glicoproteína espicular S. Como no podía ser de otra forma, su imagen con criomicroscopia electrónica se ha logrado en un tiempo récord. El equipo liderado por Jason S. McLellan, Univ. Texas en Austin (EEUU), la ha publicado en la revista Science. Gracias a esta imagen con una resolución de 3.5 Å se confirma que esta proteína S se acopla a la proteína ACE2 de las células humanas con mayor afinidad que la del coronavirus SARS-CoV. La proteína S es la diana de los anticuerpos que nos inmunizan. Su estructura 3D permite entender por qué los anticuerpos monoclonales publicados contra el SARS-CoV no son afectivos contra SARS-CoV-2. Sin lugar a dudas, ayudará a acelerar el desarrollo de vacunas y terapias contra la infección COVID-19.
La proteína S es un trímero formado por tres péptidos, cada uno con dos subunidades S1 y S2. La subunidad S1 actúa como una bisagra con dos conformaciones llamadas “abajo” (RBD down) y “arriba” (RBD up). La imagen por criomicroscopia electrónica muestra que solo uno de los péptidos está en estado “arriba”, estando los otros dos en estado “abajo”. La unión al receptor celular se realiza en la configuración “arriba”. Tras la unión se escinden los tres péptidos de la proteína S por el punto S1/S2; luego se produce una segunda escisión por el punto S2′, que despliega el péptido de fusión (FP) clave en la unión entre las membranas. La conformación tridimensional observada indica que la fusión entre el virus y el huésped es muy similar a la documentada en otros coronavirus (sobre todo para el coronavirus de la hepatitis murina, M-CoV, y para el SARS-CoV).
[PS 25 feb 2020] Se ha publicado el preprint de una segunda reconstrucción 3D de la proteína S del SARS-CoV-2 obtenida mediante criomicroscopia electrónica, obtenida por el equipo de David Veesler (Univ. Washington, Seattle, USA). Un artículo muy interesante que complementa al publicado en Science. Los interesados disfrutarán con Alexandra C. Walls, Young-Jun Park, …, David Veesler, “Structure, function and antigenicity of the SARS-CoV-2 spike glycoprotein,” bioRxiv preprint 956581 (20 Feb 2020), doi:[/PS]
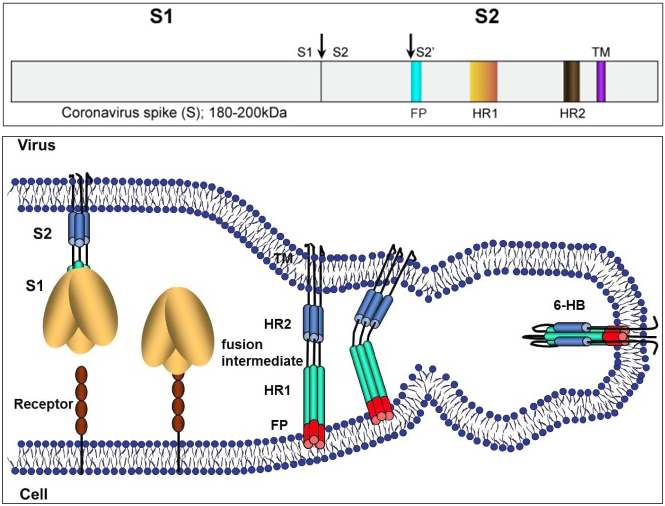
La proteína S (espicular) es una proteína trimérica transmembrana de tipo I con entre 1160 y 1400 aminoácidos, según el tipo de coronavirus. Esta proteína que forma la «corona» de los coronavirus está constituida por tres pépticos repetidos (es un trímero) y está muy glicosilada (contiene entre 21 y 35 sitios donde se adicionan carbohidratos), lo que facilita su unión a azúcares y proteínas. Cada péptido está formado por dos dominios llamados S1 y S2. En los gammacoronavirus y en algunos betacoronavirus se produce una escisión de las subunidades S1 y S2 durante la fusión entre las membranas, mientras que en los alfacoronavirus y en algunos betacoronavirus no se produce dicha escisión. SARS-CoV-2 es un betacoronavirus en el que se produce la escisión.
El dominio S1 tiene a su vez dos subdominios, uno N-terminal (NTD), que finaliza con un aminoácido que posee un grupo amino libre (-NH2), y otro C-terminal (CTD), que termina en un grupo carboxilo (-COOH); ambos se acoplan al receptor ACE2 de la célula huésped, luego son dominios de unión al receptor (RBD). El dominio S2 es de tipo C-terminal y está muy conservado entre todos los coronavirus, que se diferencian mucho más en la subunidad S1. El dominio S2 contiene dos regiones, HR1 y HR2, en las que se repiten grupos de siete aminoácidos (llamados heptads), en la forma abcdefg, conteniendo a y d residuos hidrófugos que participan en la fusión entre las membranas. Los dominios HR1 y HR2 son dianas terapéuticas, pues se conocen fármacos que inhiben su acción, evitando o dificultando la fusión.
La infección de células epiteliales de las vías respiratorias está orquestada por la proteína S del virus. La figura ilustra los pasos generales del proceso de fusión. Primero, el dominio S1 reconoce y se acopla al receptor de la célula huésped. Segundo, se produce una primera escisión de los dominios S1 y S2, y una segunda escisión en el punto S2′; esta última permite que se active el péptido de fusión (FP) que conecta las membranas del huésped y el virus (esta fase se llama etapa intermedia de fusión, o fusion-intermediate stage). Y tercero, la región entre HR1 y HR2 se reconforma (se dobla) dando lugar a un heptámero (6-HB) que une ambas membranas permitiendo la entrada del virus.
La proteína S de los coronavirus es clave en el desarrollo de vacunas (antígenos que induzcan una respuesta inmune a la presencia del dominio S1) y para el desarrollo de antivirales (inhibidores de algunas de las etapas de la fusión entre membranas, normalmente atacando regiones concretas del dominio S2). Conocer la estructura tridimensional de la proteína S es fundamental para combatir la epidemia de COVID-19.
Sist endret: lørdag, 5. juni 2021, 13:40